Electricidad y CRISPR utilizados para escribir datos en el ADN bacteriano
En los últimos años, los investigadores han utilizado el ADN para codificar todo, desde un sistema operativo a malware. En lugar de ser una curiosidad tecnológica, estos esfuerzos fueron intentos serios de aprovechar las propiedades del ADN para el almacenamiento de datos a largo plazo. El ADN puede permanecer químicamente estable durante cientos de miles de años, y es poco probable que perdamos la tecnología para leerlo, algo que no se puede decir sobre cosas como unidades ZIP y discos MO.
Pero hasta ahora, escribir datos en el ADN ha implicado convertir los datos en una secuencia de bases en una computadora y luego ordenar esa secuencia desde algún lugar que opere un sintetizador químico; los seres vivos en realidad no entran en la imagen. Pero por separado, un grupo de investigadores había estado descubriendo cómo registrar eventos biológicos modificando el ADN de una célula, lo que les permitió leer la historia de la célula. Un grupo de la Universidad de Columbia ha descubierto cómo fusionar los dos esfuerzos y escribir datos en el ADN utilizando diferencias de voltaje aplicadas a bacterias vivas.
CRISPR y almacenamiento de datos
El sistema CRISPR se ha desarrollado como una forma de editar genes o eliminarlos del ADN por completo. Pero el sistema llamó primero la atención de los biólogos porque insertaba nuevas secuencias en el ADN. Para todos los detalles, consulte nuestra cobertura Nobel, pero por ahora, solo sepa que parte del sistema CRISPR implica identificar el ADN de los virus e insertar copias del mismo en el genoma bacteriano para reconocerlo en caso de que el virus vuelva a aparecer.
El grupo de Columbia ha descubierto cómo usar esto para registrar recuerdos en bacterias. Digamos que tiene un proceso que activa genes en respuesta a una sustancia química específica, como un azúcar. Los investigadores desviaron esto para activar también un sistema que hace copias de una pieza circular de ADN llamada plásmido. Una vez que el número de copias fue alto, activaron el sistema CRISPR. Dadas las circunstancias, lo más probable es que se inserte una copia del ADN plasmídico en el genoma. Cuando el azúcar no estaba presente, generalmente insertaba algo más.
Con este sistema, fue posible saber si una bacteria ha estado expuesta al azúcar en el pasado. No es perfecto, ya que el sistema CRISPR no siempre inserta algo cuando lo desea, pero funciona en promedio. Por lo tanto, solo tiene que secuenciar suficientes bacterias para averiguar la secuencia promedio de eventos.
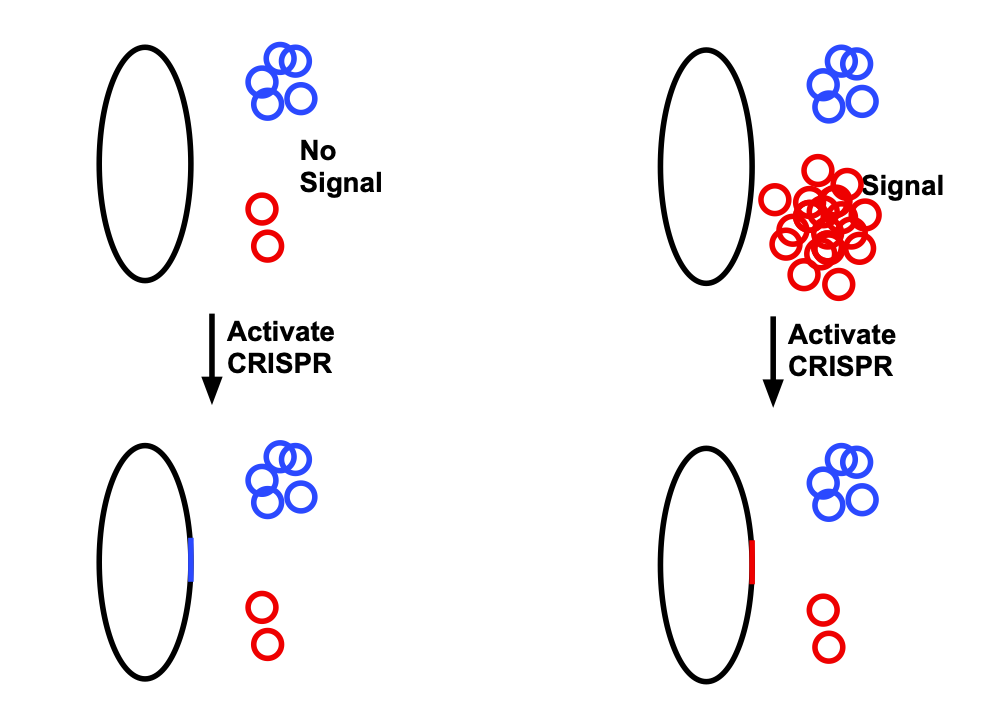
Para adaptar esto para el almacenamiento de datos, los investigadores utilizaron dos plásmidos. Uno es el mismo que se describió anteriormente: presente en niveles bajos cuando una señal específica está ausente y presente en niveles muy altos cuando la señal está presente. El segundo siempre está presente en niveles moderados. Cuando se activa CRISPR, tendía a insertar secuencias de cualquier plásmido presente en niveles más altos, como se muestra en el diagrama a continuación.
John Timmer
Por sí solo, esto solo almacena un bit. Pero el proceso se puede repetir, creando un tramo de ADN que es una serie de inserciones derivadas de los plásmidos rojo y azul, con la identidad determinada por si la señal estaba presente o no.
Dándole una sacudida
Es un sistema ordenado pero bastante alejado del tipo de cosas que normalmente asociamos con la producción de datos: la salida de una lectura o cálculo de un sensor rara vez es un azúcar o un antibiótico mezclado con un montón de bacterias. Conseguir que las bacterias respondan a una señal eléctrica resultó ser relativamente sencillo. E. coli es capaz de alterar la actividad de los genes dependiendo de si se encuentra en un entorno químico oxidante o reductor. Y los investigadores podrían alterar el medio ambiente aplicando diferencias de voltaje a una sustancia química específica en el cultivo con la bacteria.
Más específicamente, la diferencia de voltaje alteraría el estado oxidativo de un químico llamado ferrocianuro. Eso, a su vez, provocó que las bacterias alteren la actividad de los genes. Al diseñar el plásmido para que respondiera a la misma señal que estos genes, los investigadores pudieron controlar los niveles de plásmido aplicando diferentes voltajes. Y luego podrían registrar ese nivel de ese plásmido activando el sistema CRISPR en estas células.
Es bastante fácil ver cómo cada uno de los insertos de una serie podría considerarse un cero o uno, dependiendo de la identidad del inserto. Pero recuerde que este sistema no es perfecto; Con bastante frecuencia, CRISPR no inserta nada cuando está activado, lo que desplaza todos los bits siguientes. Como este proceso es aleatorio, cuanto más larga sea la serie de bits que intente codificar, más probable será que al menos uno de ellos termine siendo omitido.
Para limitar este problema, los investigadores mantuvieron sus datos en tres bits por población bacteriana. Incluso entonces, tuvieron que entrenar un algoritmo de aprendizaje supervisado para reconstruir la serie de bits más probable basándose en un promedio de las secuencias encontradas en la población. E, incluso con eso, el sistema no reconoció la serie de bits alrededor del seis por ciento de las veces. Al final, decidieron usar un bit de paridad que era la suma de los dos primeros para permitir la corrección de errores y luego editaron lotes de poblaciones en paralelo.
(Al dar a los plásmidos de cada población una etiqueta de secuencia única llamada “código de barras”, fue posible mezclar muchos de ellos en una sola población después de que se codificaron los bits y aún así desenredar todo una vez que se secuenció el ADN).
Con todo en su lugar, almacenaron y leyeron correctamente “¡Hola mundo!” Incluso pusieron la bacteria en tierra para macetas durante una semana y demostraron que pudieron recuperar el mensaje. (Guardarlos en el congelador obviamente funciona mejor). Ellos estiman que el mensaje se puede retener durante al menos 80 generaciones de bacterias.
Seamos claros: como medio de almacenamiento, en su forma actual, esto es bastante terrible. Si quisiera poner algunos datos en el ADN, sería mucho mejor que sintetizara químicamente el ADN. Pero es intrigante pensar que podríamos pasar directamente de las señales eléctricas al ADN alterado, y puede haber algunas formas de mejorar el sistema ahora que se ha establecido.
Biología química de la naturaleza, 2021. DOI: 10.1038 / s41589-020-00711-4 (Acerca de los DOI).

“Web hayranı. Tipik düşünür. İçine kapanık. Amatör iletişimci. Pop kültürü meraklısı.”
